La PCR classique
La Polymerase Chain Reaction (PCR) technique mise au point par K. Mullis en 1985. Elle consiste à amplifier in vitro, via d’une réaction enzymatique, un segment d’ADN connu en un très grand nombre de copies à partir d’une faible quantité dans le but de permettre sa détection.
La technique connaît un essor considérable à partir de la commercialisation (vers 1988) d'une ADN polymérase thermorésistante : la Taq polymérase, qui a permis une "automatisation" de la technique .
On introduit dans un même tube tous les "acteurs" de la PCR. Il s'agit de l'ADN matrice à amplifier, des amorces spécifiques du segment d'ADN cible (sens et anti-sens), de l'ADN polymérase thermostable, du mélange des 4 dNTPs constitutifs de l'ADN et du tampon qui contient notamment du MgCl2, apportant ainsi du magnésium qui est un co-facteur essentiel aux ADN polymérases.
Chaque cycle de PCR est constitué de trois étapes (dans le cas d'une PCR à 3 segments) : - la dénaturation (94°C) : séparation des doubles brins d’ADN - l'hybridation (entre 50 et 65°C) : hybridation des amorces sur l'ADN matrice - l'élongation (72°C) : synthèse du brin complémentaire
Les cycles d'amplification sont réalisés à l’aide d’un thermocycleur. C'est un appareil capable de varier très rapidement de température, notamment grâce à l'effet Peltier
Une fois l'amplification terminée, on vérifie que l'amplification est bien spécifique, en faisant migrer nos produits de PCR sur gel d'agarose (Cf. Électrophorèse).
Du DMSO peut être ajouté en additif dans le milieu réactionnel de la PCR à une concentration finale de 5% afin d'augmenter le rendement d'amplification en réduisant la formation de structures secondaires.
Dans le but de réduire au maximum les hybridations non spécifiques et donc la formation d’amplicons non souhaités, il est également possible de réaliser une Touchdown PCR. Pour cela, la température d’hybridation est placée au-dessus de la Tm des amorces puis est diminuée pendant plusieurs cycles jusqu’à atteindre la Tm des amorces. Les premiers cycles permettent d’augmenter la stringence et donc de n’amplifier que nos séquences cibles. Les cycles suivants permettent d’augmenter le rendement d’amplification en abaissant la température d‘hybridation à la Tm des amorces.
La PCR quantitative en temps réel
La PCR quantitative (qPCR) est une PCR qui se base sur le même principe que la PCR en temps réel: on suit la cinétique de formation d'amplicons en temps réel via un signal fluorescent.
Il existe deux méthodes d'analyse en qPCR : la méthode au SYBR Green I et celle utilisant des sondes Taqman.
La technologie de PCR quantitative repose sur une notion spécifique primordiale : le cycle seuil ou "cycle threshold" (Ct).
Le cycle seuil correspond au nombre de cycles à partir duquel la quantité de produit formé est distinct du "bruit de fond". Ce Ct marque le début de la phase exponentielle d’amplification (= phase reproductible non limitante).
Le Ct est inversement proportionnel au logarithme de base 2 du nombre de copies initiales présentes de la cible dans le milieu réactionnel : plus le Ct d’un échantillon est petit, plus le nombre de copies initiales de la cible est important.
Le SYBR Green est un intercalant de l'ADN non spécifique qui s'insère dans le petit sillon de l'ADN double brin.
Cette molécule émet très peu de fluorescence à l’état libre, tandis qu'une fois incorporée dans l'ADN il émet fortement à 520 nm. Cette capacité de se fixer à l’ADN bicaténaire conduit à la limite de cette méthode car il y a un risque de détecter des dimères d’amorces ou une amplification non spécifique.
La fluorescence est mesurée à la fin de chaque étape d’élongation de chaque cycle, l’augmentation de fluorescence traduit donc la formation d’amplicons.
Analyse de la spécificité des amorces
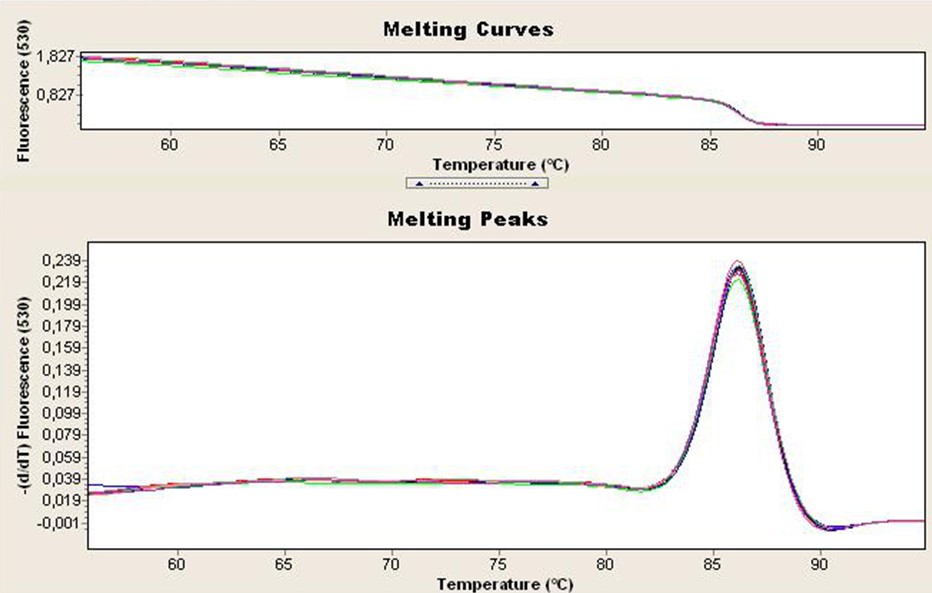
Les différents couples d’amorces utilisés sont testés en qPCR afin de vérifier leurs spécificités. La courbe de fusion permet de vérifier qu’il n’y a qu’un seul produit amplifié et donc que les couples d’amorces utilisés sont bien spécifiques.
Pour cela, la température est élevée à 95°C afin que la totalité des brins d’ADN soient sous forme monocaténaire. On abaisse ensuite la température à la température d’hybridation des amorces. Cela permet aux amplicons de s’apparier et aux éventuels dimères d’amorces de se former. Enfin, on augmente lentement la température jusqu’à 95°C en mesurant la fluorescence de manière continue. Lorsqu’on atteint la température de fusion des hybrides formés, on obtient la moitié sous forme bicaténaire et l’autre moitié sous forme monocaténaire, ce qui provoque une chute du signal de fluorescence.
Chaque produit d’amplification a une température de fusion qui dépend de la composition et de la longueur de sa séquence nucléotidique.
En traçant la dérivé primaire de cette courbe, on obtient une courbe comme celle présentée ci-dessous. La présence d’un pic unique et symétrique signifie qu’on a obtenu un seul type d’amplicon et donc que le couple d’amorces utilisés est spécifique du fragment recherché.
Il existe deux types de quantifications :
- la quantification absolue qui s’appuie sur des gammes étalons de dilutions standard de concentration connue permettant de déterminer le nombre de copies initiales de la séquence cible.
- la quantification relative qui permet de comparer l’expression d’un gène cible dans une condition particulière par rapport à une condition témoin. On s'appuie pour cela, sur un gène de référence dont l'expression ne varie pas selon le stade de développement ou le traitement réalisé chez le tissu étudié.
Un gène de référence (ou « standard ») est un gène dont l’expression ne varie pas quel que soit le stade de développement ou le traitement réalisé chez le tissu étudié. Il permet de normaliser les éventuels biais pouvant provenir des variations dans les erreurs de pipetage et des variations d’efficacité lors de la transcription inverse.
On choisit généralement des gènes codant des protéines ayant des fonctions indispensables au bon fonctionnement cellulaire (structure du cytosquelette, protéine de la sous-unité ribosomale, protéine du métabolisme,..)
